


 النبات
النبات
 الحيوان
الحيوان
 الأحياء المجهرية
الأحياء المجهرية
 علم الأمراض
علم الأمراض
 التقانة الإحيائية
التقانة الإحيائية
 التقنية الحيوية المكروبية
التقنية الحيوية المكروبية
 التقنية الحياتية النانوية
التقنية الحياتية النانوية
 علم الأجنة
علم الأجنة
 الأحياء الجزيئي
الأحياء الجزيئي
 علم وظائف الأعضاء
علم وظائف الأعضاء
 الغدد
الغدد
 المضادات الحيوية
المضادات الحيوية|
Read More
Date: 7-10-2021
Date: 28-12-2021
Date: 5-10-2021
|
Prokaryotic DNA Replication : Chain elongation
Prokaryotic (and eukaryotic) DNA pols elongate a new DNA strand by adding deoxyribonucleotides, one at a time, to the 3′-end of the growing chain (see Fig. 1). The sequence of nucleotides that are added is dictated by the base sequence of the template strand with which the incoming nucleotides are paired.
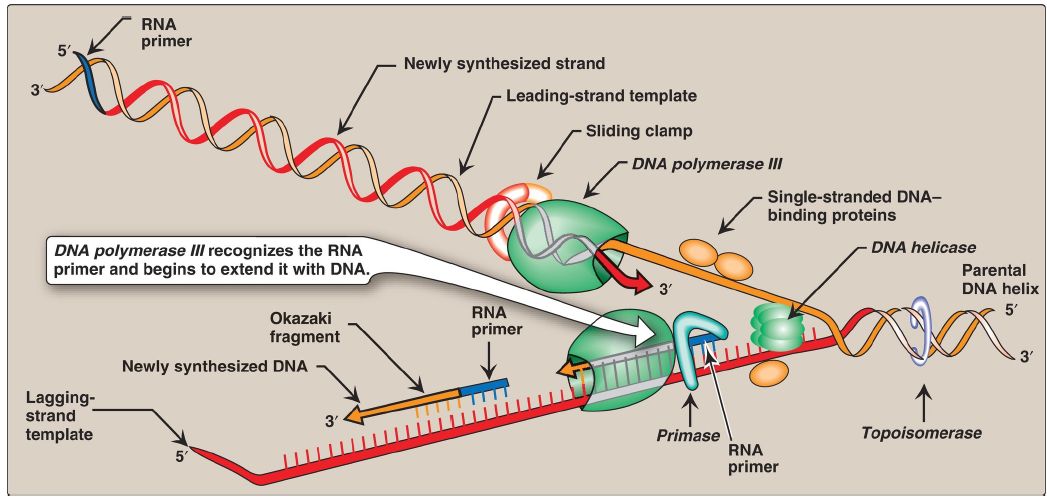 Figure 1: Elongation of the leading and lagging strands. [Note: The DNA sliding clamp is not shown for the lagging strand.]
Figure 1: Elongation of the leading and lagging strands. [Note: The DNA sliding clamp is not shown for the lagging strand.]
1. DNA polymerase III: DNA chain elongation is catalyzed by the multisubunit enzyme, DNA pol III. Using the 3′-hydroxyl group of the RNA primer as the acceptor of the first deoxyribonucleotide, DNA pol III begins to add nucleotides along the single-stranded template that specifies the sequence of bases in the newly synthesized chain. DNA pol III is a highly processive enzyme (that is, it remains bound to the template strand as it moves along and does not diffuse away and then rebind before adding each new nucleotide). The processivity of DNA pol III is the result of the β subunits of the holoenzyme forming a ring that encircles and moves along the template strand of the DNA, thus serving as a sliding DNA clamp. [Note: Clamp formation is facilitated by a protein complex, the clamp loader, and ATP hydrolysis.] The new (daughter) strand grows in the 5′→3′ direction, antiparallel to the parental strand (see Fig. 1). The nucleotide substrates are 5′-deoxyribonucleoside triphosphates. Pyrophosphate (PPi) is released when each new deoxynucleoside monophosphate is added to the free 3′-hydroxyl group of the growing chain through a 3′→5′-phosphodiester bond . Hydrolysis of PPi to 2 Pi by pyrophosphatase means that a total of two high-energy bonds are used to drive the addition of each deoxynucleotide.
The production of PPi with subsequent hydrolysis to 2 Pi is a common theme in biochemistry. Removal of the PPi product drives a reaction in the forward direction, making it essentially irreversible.
All four substrates (deoxyadenosine triphosphate [dATP], deoxythymidine triphosphate [dTTP], deoxycytidine triphosphate [dCTP], and deoxyguanosine triphosphate [dGTP]) must be present for DNA elongation to occur. If one of the four is in short supply, DNA
synthesis stops when that nucleotide is depleted.
2. Proofreading newly synthesized DNA: It is highly important for the survival of an organism that the nucleotide sequence of DNA be replicated with as few errors as possible. Misreading of the template sequence could result in deleterious, perhaps lethal, mutations. To insure replication fidelity, DNA pol III has a proofreading activity (3′→5′ exonuclease, Fig. 30.17) in addition to its 5′→3′ polymerase activity. As each nucleotide is added to the chain, DNA pol III checks to make certain the base of the newly added nucleotide is, in fact, the complement of the base on the template strand. If it is not, the 3′→5′ exonuclease activity removes the error in the direction opposite to polymerization.
[Note: Because the enzyme requires an improperly base-paired 3′-hydroxy terminus, it does not degrade correctly paired nucleotide sequences.] For example, if the template base is C and the enzyme inserts an A instead of a G into the new chain, the 3′→5′ exonuclease activity hydrolytically removes the misplaced nucleotide. The 5′→3′ polymerase activity then replaces it with the correct nucleotide containing G (see Fig. 2). [Note: The 5′→3′ polymerase and 3′→5′ exonuclease domains are located on different subunits of DNA pol III.]

Figure 2: 3′→5′ Exonuclease activity enables DNA polymerase III to proofread the newly synthesized DNA strand.

|
|
|
|
إدارة الغذاء والدواء الأميركية تقرّ عقارا جديدا للألزهايمر
|
|
|
|
|
|
|
شراء وقود الطائرات المستدام.. "الدفع" من جيب المسافر
|
|
|
|
|
|
|
بالصور: الامين العام للعتبة الحسينية المقدسة يجري جولة ميدانية للوقوف على آخر الاستعدادات الخاصة بمراسيم تبديل راية الإمام الحسين (ع)
|
|
|