


 النبات
النبات
 الحيوان
الحيوان
 الأحياء المجهرية
الأحياء المجهرية
 علم الأمراض
علم الأمراض
 التقانة الإحيائية
التقانة الإحيائية
 التقنية الحيوية المكروبية
التقنية الحيوية المكروبية
 التقنية الحياتية النانوية
التقنية الحياتية النانوية
 علم الأجنة
علم الأجنة
 الأحياء الجزيئي
الأحياء الجزيئي
 علم وظائف الأعضاء
علم وظائف الأعضاء
 الغدد
الغدد
 المضادات الحيوية
المضادات الحيوية|
Read More
Date: 21-12-2015
Date: 25-12-2015
Date: 28-5-2021
|
Hybridisation Techniques
The principle of complementarity, according to which A base pairs with T and C base pairs with G in a nucleic acid duplex, is the core principle of heredity, all hybridisation techniques and the PCR alike. The A–T base pairing involves two and the G–C base pairing involves three hydrogen bonds. Therefore, the relative content of A–T and G–C base pairs contributes strongly to the Tm of the nucleic acid duplex. The higher the G–C content, the higher is the Tm of the nucleic acid duplex. Different types of hybridisations were among the first techniques attempted in the laboratory detection of nucleic acids. In general, it turned out that the sensitivity of hybridisation techniques was not sufficient for routine detection of infectious agents directly in patient samples. The invention of nucleic acid amplification techniques therefore represented a breakthrough in nucleic acid-based laboratory diagnosis.
However, in some cases the sensitivity of hybridisation was promising and encouraged further development of methods to detec nucleic acids, in particular for bacteria containing multicopy plasmids or repeated genomic sequences.
1- Sandwich Hybridisation
In the classical hybridisation techniques, the nucleic acid target was usually fixed on a solid phase (nitrocellulose, nylon membrane, glass slide) and detected by a labelled probe added in the hybridisation buffer to the solid phase. These techniques, such as dot blots, colony blots, Southern blots, Northern blots and in situ hybridisation, usually either lacked sufficient sensitivity due to background hybridisation or were too laborious for the routine setting. The sandwich hybridisation (Figure 1) technique turned out, however, to have sufficiently low background and strong enough signal to be employed for some routine laboratory purposes. In this technique, the capture probe is fixed to the solid phase and available to the target in the hybridisation buffer. Following annealing between the capture probe and the specific target, unbound material is removed by washing and a new hybridisation buffer containing a detection probe is added. The detection probe is typically labelled with either biotin or an enzyme and is designed to bind to a different region of the target nucleic acid than the capture probe. The detection probe will therefore stick to the capture probe and the solid phase only if bridged by the specific target. Following a wash, retained detection probe can be visualised by addition of a colourless substrate that will change to a coloured product by the enzyme conjugated to the detection probe or by a streptavidin–enzyme complex in the case of a biotin-labelled detection probe. The solid phase may be a magnetic particle or the bottom of a 96-well plate to facilitate automation. The sandwich hybridisation technique is used in commercial tests for the detection of human papillomaviruses (HPVs) in samples from the female cervix (Digene) and in the visualisation step of conventional PCR assays (e.g. several Roche assays). The principle is also utilised in line probe assays.
2- Line Probe Assays
In line probe assays (Figure 2), capture probes are printed in parallel bands (lines) on a membrane. Line probe assays have been developed to distinguish strains or genotypes of infectious agents. Each band or lane contains capture probes that differ only in nucleotides that are characteristic for each strain or genotype. The target nucleic acid is typically first amplified by PCR utilizing biotin-labelled primers before addition to the line probe membrane in a hybridisation buffer. Following a wash, a streptavidin-enzyme conjugate is added and allowed to bind to biotin.
Following another wash a colourless substrate is added and will be changed by the enzyme to a precipitable coloured product. The line or band corresponding to one specific genotype will then become coloured (Figure 2).
Commercial line probe assays are used for the detection of genotypes of HPV and hepatitis C virus (HCV), strains of atypical mycobacteria and for detection of HTLV-1 (human T-cell lymphotropic virus).
3- Peptide Nucleic Acid Fluorescent In situ Hybridisation (PNA-FISH)
PNA-FISH is one type of in situ hybridisation utilising peptide nucleic acids as hybridisation probes. Peptide nucleic acids exhibit rapid hybridisation kinetics and, when coupled to fluorescent reporter molecules, offer a rapid and sensitive verification of the infectious agent in positive blood cultures. Once a blood culture turns positive, a Gram stain is performed and, based on the results, the appropriate PNA-FISH tests are selected. Identification results are available within just a few hours and can be reported to the attending physician (Figure 3).
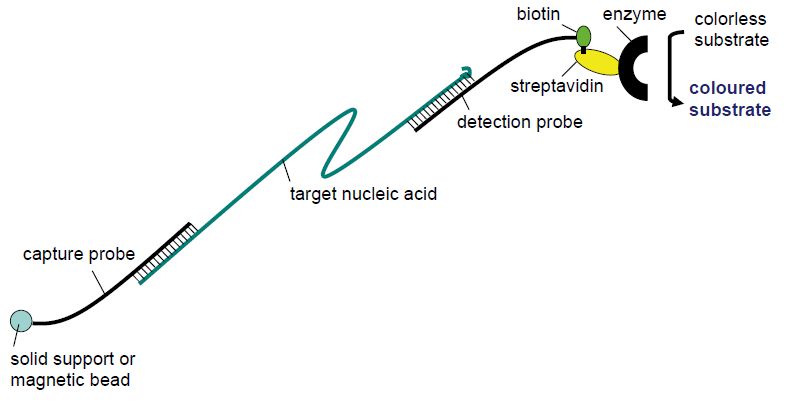
Figure 1: Principle of sandwich hybridisation
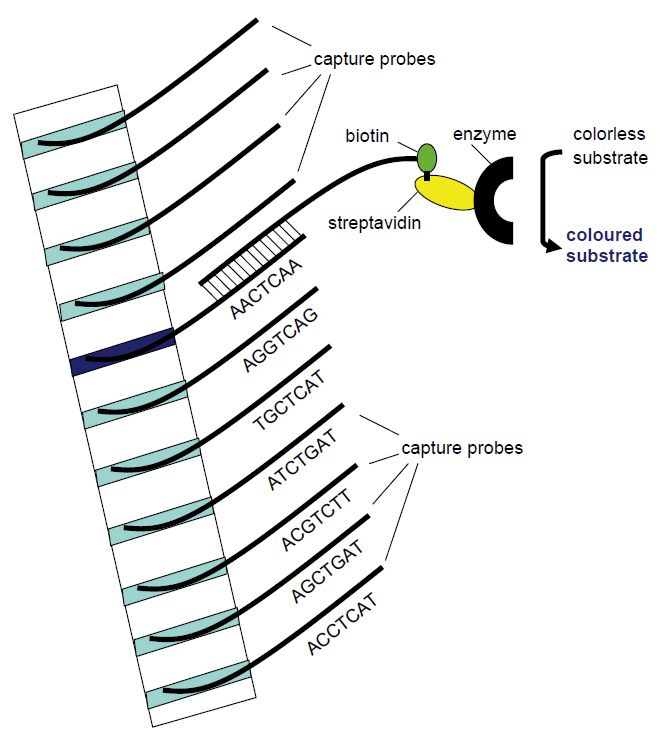
Figure 2: Principle of the line probe assay. Different capture probe sequences are fixed in different bands in a membrane strip. The amplification product will hybridise with the capture probe of complementary sequence. Utilising biotin-labelled PCR primers, the position of the complementary capture probe can be visualised as a coloured band following addition of streptavidin-bound enzyme that will change a non-coloured substrate to a coloured product that precipitates in the band.

Figure 3: Schematics of PNA-FISH. Image provided by AdvanDx (Woburn, MA, USA).

|
|
|
|
التوتر والسرطان.. علماء يحذرون من "صلة خطيرة"
|
|
|
|
|
|
|
مرآة السيارة: مدى دقة عكسها للصورة الصحيحة
|
|
|
|
|
|
|
نحو شراكة وطنية متكاملة.. الأمين العام للعتبة الحسينية يبحث مع وكيل وزارة الخارجية آفاق التعاون المؤسسي
|
|
|