النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الميكروبية

مواضيع عامة في المضادات الميكروبية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
Recombination-Repair of Double-Strand Breaks in Eukaryotes
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المصدر:
LEWIN’S GENES XII
الجزء والصفحة:
21-4-2021
2040
Recombination-Repair of Double-Strand Breaks in Eukaryotes
KEY CONCEPTS
- The yeast RAD mutations, identified by radiationsensitive phenotypes, are in genes that encode repair proteins.
- The RAD52 group of genes is required for recombination-repair.
- The MRX (yeast) or MRN (mammals) complex is required to form a single-stranded overhang at each DNA end.
- The RecA homolog Rad51 forms a nucleoprotein filament on the single-stranded regions, assisted by Rad52 and Rad55/57.
- Rad54 and Rdh54/Rad54B are involved in homology search and strand invasion.
When a replication fork encounters a lesion in a single stand, it can result in the formation of a DSB. DSBs are one of the most severe types of DNA damage that can occur, particularly in eukaryotes. If a DSB on a linear chromosome is not repaired, the portion of the chromosome lacking a centromere will not be segregated at the next cell division. In addition to their occurrence during replication, DSBs can be generated in a number of other ways, including ionizing radiation, oxygen radicals generated by cellular metabolism, action of endonucleases, attempted excision repair of clustered lesions, or encountering a nick during replication. Four pathways of DSB repair have been identified: homology-directed recombination-repair (HRR; the only error-free pathway), singlestrand annealing (SSA), alternative or microhomology-mediated end joining (alt-EJ), and nonhomologous end joining (NHEJ).
The ideal mechanism for repairing DSBs is to use HRR, as this ensures that no critical genetic information is lost due to sequence loss at the breakpoint. HRR is used predominantly during the S and G2 phases of the cell cycle, when a sister chromatid is available to provide the homologous donor sequence.
Several of the genes required for recombination-repair in eukaryotes have already been discussed in the context of homologous recombination . Many eukaryotic repair genes are named RAD genes; they were initially characterized genetically in yeast by virtue of their sensitivity to radiation. Three general groups of repair genes have been identified in the yeast S. cerevisiae: the RAD3 group (involved in excision repair), the RAD6 group (required for postreplication repair), and the RAD52 group (concerned with recombination-like mechanisms). Homologs of these genes are present in multicellular eukaryotes as well. The RAD52 group plays essential roles in homologous recombination and includes a large number of genes, including RAD50, RAD51, RAD54, RAD55, RAD57, and RAD59. These Rad proteins are all required at different stages of repair of a DSB.
After a break is detected and damage signaling occurs, a stage known as “end clipping” occurs in which the nucleases Mre11 and CtIP trim about 20 nucleotides to generate short single-stranded tails with 3′–OH overhangs. This single-stranded DNA serves to activate a DNA damage checkpoint, stopping cell division until the damage can be repaired. If short sequences in these overhangs are able to base pair (microhomologies), then the alt-EJ pathway can take over, trimming and ligating the ends, with some loss of sequence. Alternatively, as occurs during meiotic recombination, the Mre11/Rad50/Xbs1 (MRX) complex (MRN in mammals) shown in FIGURE 1, works in concert with exonucleases and helicases to further resect the ends of the DSB to generate long single-stranded tails. Extensive homology in these longer tails can engage the SSA pathway, which results in large deletions. The factors that control which pathway dominates at any repair event are complex and still not well understood.
In the highly accurate HRR pathway, the RecA homolog Rad51 binds to the single-stranded DNA to form a nucleoprotein filament, which is used for strand invasion of a homologous sequence. Rad52 and the Rad55/57 complex are required to form a stable Rad51 filament, and Rad54 and its homolog Rdh54 (Rad54B in mammals) assist in the search for homologous donor DNA and subsequent strand invasion. Rad54 and Rdh54 are members of the SWI2/SNF2 superfamily of chromatin-remodeling enzymes and may be necessary for reconfiguring chromatin structure at both the damage site and at the donor DNA. Following repair synthesis, the resulting structure (which resembles a Holliday junction) is resolved .
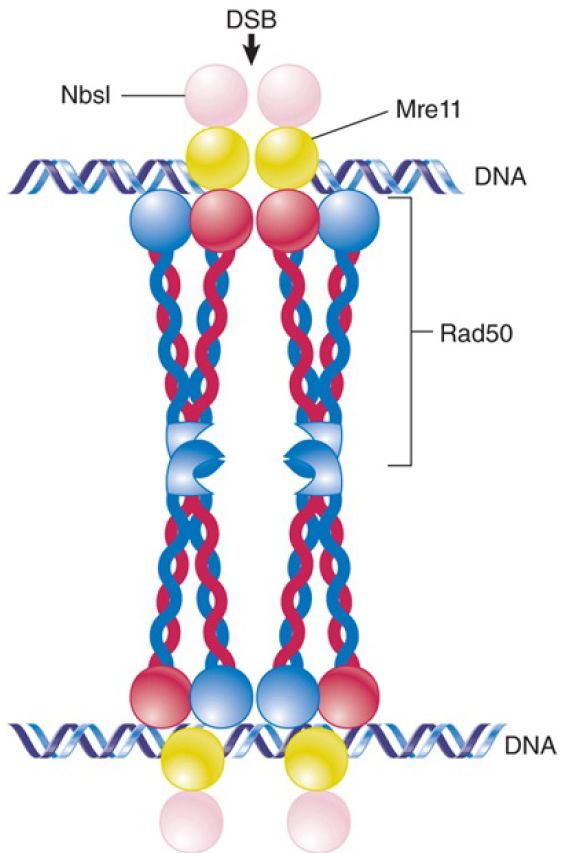
FIGURE 1.The MRN complex, required for 5’-end resection, also serves as a DNA bridge to prevent broken ends from separating. The “head” region of Rad50, bound to Mre11, binds DNA, while the extensive coiled coil region of Rad50 ends with a “zinc hook” that mediates interaction with another MRN complex. The precise position of Nbs1 within the complex is unknown, but it interacts directly with Mre11.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة

الآخبار الصحية















 قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة
قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة "المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة
"المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة (نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)
(نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)