النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الميكروبية

مواضيع عامة في المضادات الميكروبية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
A Model for Enzyme Movement Is Suggested by the Crystal Structure
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المصدر:
LEWIN’S GENES XII
الجزء والصفحة:
5-5-2021
2050
A Model for Enzyme Movement Is Suggested by the Crystal Structure
KEY CONCEPTS
- DNA moves through a channel in RNA polymerase and makes a sharp turn at the active site.
- Changes in the conformations of certain flexible modules within the enzyme control the entry of nucleotides to the active site.
- Translocation proceeds by a Brownian ratchet mechanism.
As a result of the crystal structures of the bacterial and yeast enzymes in complex with NTPs and/or with DNA, we now have considerable information about the structure and function of RNA polymerase during elongation. Bacterial RNA polymerase has overall dimensions of approximately 90 × 95 × 160 Å, and the archaeal and eukaryotic RNA polymerases are only slightly larger, primarily from additional stretches of amino acids and/or extra subunits situated on the periphery of the enzyme. Nevertheless, the core enzymes share not only a common structure, in which there is a “channel” about 25 Å wide that accommodates the DNA, but a common mechanism for nucleotide addition.
A model of this channel in bacterial RNA polymerase is illustrated in FIGURE 1. The groove holds about 17 bp of DNA. In conjunction with the approximately 13 nucleotides of DNA accommodated by the enzyme’s active site region, this accounts for the approximately 30- to 35-nucleotide protected region observed in footprints of the elongation complex. The groove is lined with positive charges, enabling it to interact with the negatively charged phosphate groups of DNA. The catalytic site is formed by a cleft between the two large subunits that grasp DNA downstream in its “jaws” as it enters the RNA polymerase. RNA polymerase surrounds the DNA, and a catalytic Mg ion is found at the active site. The DNA is held in position by the downstream clamp, another name for one of the jaws. FIGURE 2 illustrates the 90° turn that the DNA takes at the entrance to the active site because of an adjacent wall of protein. The length of the RNA hybrid is limited by another protein obstruction, called the lid. Nucleotides are thought to enter the active site from below, via the secondary channel (called the pore in yeast RNA polymerase). The transcription bubble includes 8 to 9 bp of DNA–RNA hybrid. The lid separates the DNA and RNA bases at one end of the hybrid (see Figure 17.24), and the DNA base on the template strand at the other end of the hybrid is flipped out to allow pairing with the incoming NTP.
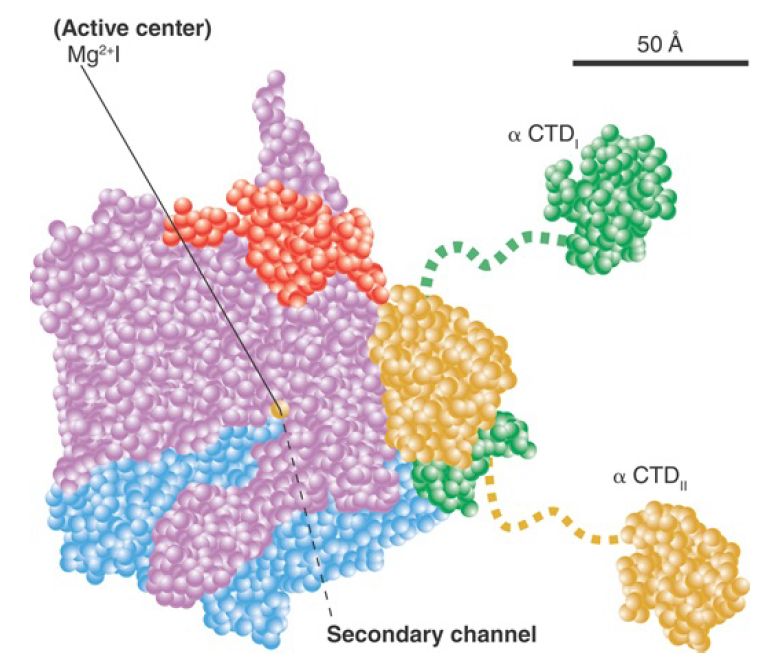
FIGURE 1. The A model showing the structure of RNA polymerase through the main channel. Subunits are color-coded as follows: β′, pink; β, cyan; αI, green; αII, yellow; ω, red.
Data from K. M. Geszvain and R. Landick (ed. N. P. Higgins). The Bacterial Chromosome. American Society for Microbiology, 2004.
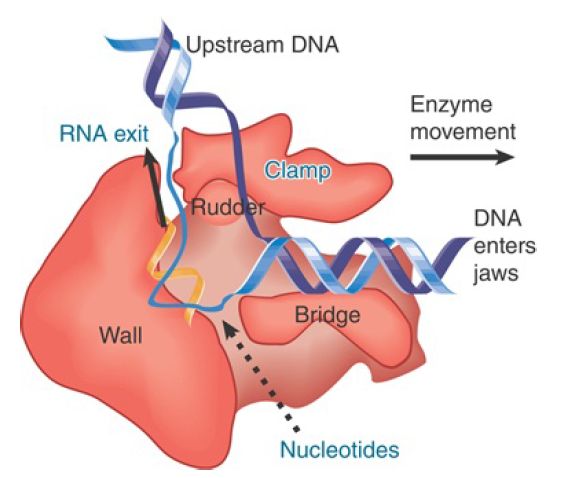
FIGURE 2. DNA is forced to make a turn at the active site by a wall of protein. Nucleotides may enter the active site through a pore in the protein.
Once DNA has been melted, the trajectory of the individual strands within the enzyme is no longer constrained by the rigidity of the double helix, allowing DNA to make its 90° turn at the active site. Furthermore, a large conformational change occurs in the enzyme itself involving the downstream clamp.
One of the dilemmas of any nucleic acid polymerase is that the enzyme must make tight contacts with the nucleic acid substrate and product, but then must break these contacts and remake them with each cycle of nucleotide addition. Consider the situation illustrated in FIGURE 3. A polymerase makes a series of specific contacts with the bases at particular positions. For example, contact “1” is made with the base at the end of the growing chain and contact “2” is made with the base in the template strand that is complementary to the next base to be added. Note, however, that the bases that occupy these locations in the nucleic acid chains change every time a nucleotide is added!
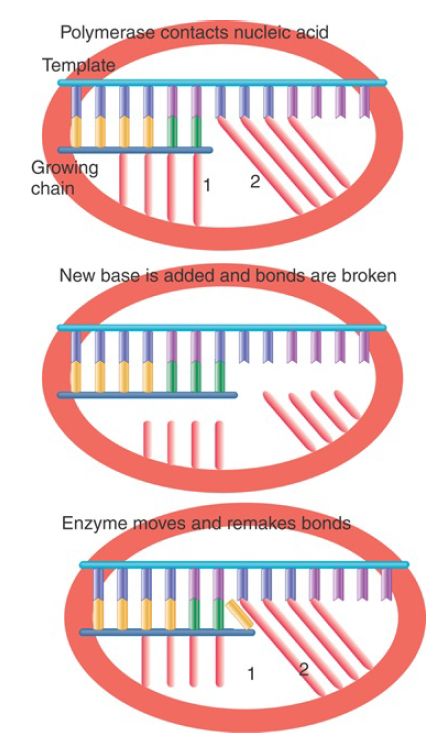
FIGURE 3. Movement of a nucleic acid polymerase requires breaking and remaking bonds to the nucleotides at fixed positions relative to the enzyme structure. The nucleotides in these positions change each time the enzyme moves a base along the template.
The top and bottom panels of the figure show the same situation: A base is about to be added to the growing chain. The difference is that the growing chain has been extended by one base in the bottom panel. The geometry of both complexes is exactly the same, but contacts “1” and “2” in the bottom panel are made to bases in the nucleic acid chains that are located one position farther along the chain. The middle panel shows that this must mean that, after the base is added, and before the enzyme moves relative to the nucleic acid, the contacts made to specific positions must be broken so that they can be remade to bases that occupy those positions after the movement.
RNA polymerase crystal structures provide considerable insight into how the enzyme retains contact with its substrate while breaking and remaking bonds in the process of the nucleotide addition cycle and undergoing translocation by a Brownian ratchet mechanism. Random fluctuations occur and are locked into the correct position by the binding of a nucleoside triphosphate. The energy from binding the correct substrate stabilizes the active conformation and suppresses backtracking. A flexible module called the trigger loop appears to be unfolded before nucleotide addition, but becomes folded once the NTP enters the active site. Once bond formation and translocation of the enzyme to the next position are complete, the trigger loop unfolds again, ready for the next cycle. Thus, a structural change in the trigger loop coordinates the sequence of events in catalysis.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة

الآخبار الصحية















 قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة
قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة "المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة
"المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة (نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)
(نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)