النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الميكروبية

مواضيع عامة في المضادات الميكروبية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
Insertion Sequences Are Simple Transposition Modules
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المصدر:
LEWIN’S GENES XII
الجزء والصفحة:
22-4-2021
3545
Insertion Sequences Are Simple Transposition Modules
KEY CONCEPTS
- An insertion sequence is a transposon that encodes the enzyme(s) needed for transposition flanked by short inverted terminal repeats.
- The target site at which an insertion sequence is inserted is duplicated during the insertion process to form two repeats in direct orientation at the ends of the transposon.
- The length of the direct repeat is 5 to 9 bp and is characteristic for any particular insertion sequence.
Transposable elements were first identified at the molecular level in the form of spontaneous insertions in bacterial operons. Such an insertion prevents transcription and/or translation of the gene in which it is inserted. Many different types of transposable elements have now been characterized in both prokaryotes and eukaryotes (they are far more abundant in the latter), but the basic principles and biochemistry of elements first described in bacteria apply to DNA-type elements in many species.
The simplest bacterial transposons are called insertion sequence (IS) elements (reflecting the way in which they were detected). Each type is given the prefix “IS,” followed by a number that identifies the type. (The original classes were numbered IS1 to IS4; later classes have numbers reflecting the history of their isolation, but not corresponding to the more than 700 elements so far identified!)
The IS elements are normal constituents of bacterial chromosomes and plasmids. A standard strain of Escherichia coli is likely to contain several (fewer than 10) copies of any one of the more common IS elements. To describe an insertion into a particular site, a double colon is used; thus λ::IS1 describes an IS1 element inserted into phage lambda. Most IS elements insert at a variety of sites within host DNA. Some, though, show varying degrees of preference for particular hotspots.
The IS elements are autonomous units, each of which encodes only the proteins needed to sponsor its own transposition. Each IS element is different in sequence, but there are some common features in organization. The structure of a generic transposon before and after insertion at a target site is illustrated in FIGURE 1, which also summarizes the details of some common IS elements.
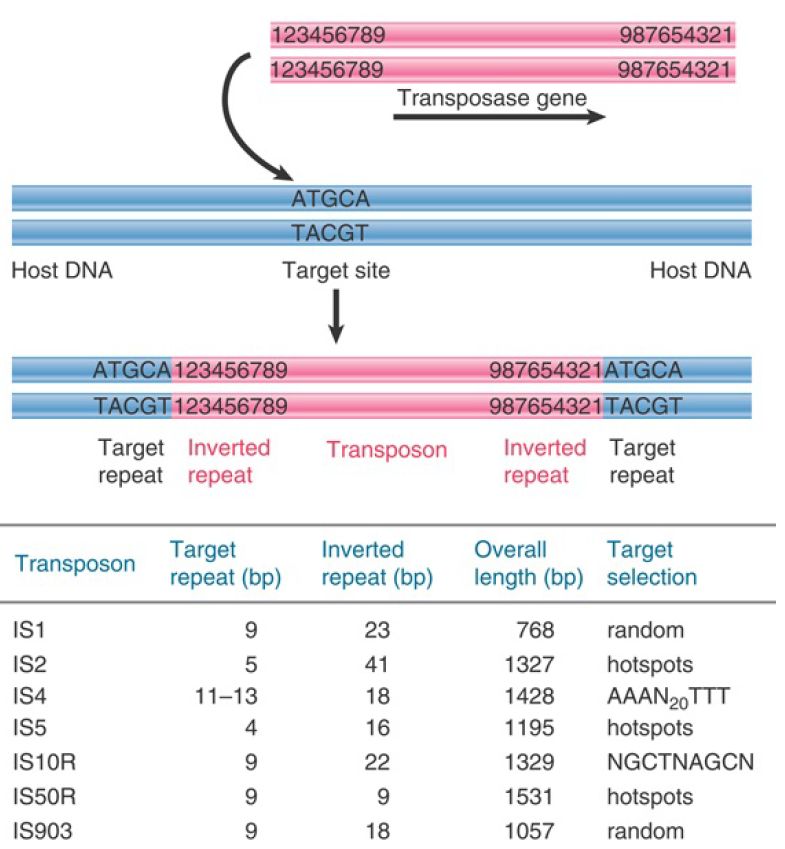
FIGURE 1. IS elements have inverted terminal repeats and generate direct repeats of flanking DNA at the target site. In this example, the target is a 5-bp sequence. The ends of the transposon consist of inverted repeats of 9 bp, where the numbers 1 through 9 indicate a sequence of base pairs.
An IS element ends in short inverted terminal repeats; usually the two copies of the repeat are closely related rather than identical. As illustrated in Figure 15.3, the presence of the inverted terminal repeats means that the same sequence is encountered proceeding toward the element from the flanking DNA on either side of it. When an IS element transposes, a sequence of host DNA at the site of insertion is duplicated. The nature of the duplication is revealed by comparing the sequence of the target site before and after an insertion has occurred. Figure 15.3 shows that at the site of insertion the IS DNA is always flanked by very short direct repeats. (In this context, “direct” indicates that two copies of a sequence are repeated in the same orientation, not that the repeats are adjacent.) In the original gene (prior to insertion), however, the target site has the sequence of only one of these repeats. In the figure, the target site consists of the sequence 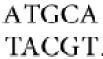 . After transposition, one copy of this sequence is present on either side of the transposon. The sequence of the direct repeat varies among individual transposition events undertaken by a transposon, but the length is constant for any particular IS element (a reflection of the mechanism of transposition).
. After transposition, one copy of this sequence is present on either side of the transposon. The sequence of the direct repeat varies among individual transposition events undertaken by a transposon, but the length is constant for any particular IS element (a reflection of the mechanism of transposition).
An IS element therefore displays a characteristic structure in which its ends are identified by the inverted terminal repeats, whereas the adjacent ends of the flanking host DNA are identified by the short direct repeats. When observed in a sequence of DNA, this type of organization is taken to be diagnostic of a transposon and suggests that the sequence originated in a transposition event.
The inverted repeats define the ends of a transposon. Recognition of the ends is common to transposition events sponsored by all types of DNA-type transposon. cis-acting mutations that prevent transposition are located in the ends, which are recognized by a protein(s) responsible for transposition. The protein is called a transposase.
Many of the IS elements contain a single, long coding region, which starts just inside the inverted repeat at one end and terminates just before or within the inverted repeat at the other end. This region encodes the transposase. Some elements have a more complex organization. IS1, for instance, has two separate reading frames; the transposase is produced by making a frameshift during translation to allow both reading frames to be used.
The frequency of transposition varies among different elements. Under most circumstances the overall rate of transposition is 10-3 to 10-4 per element per generation. Insertions in individual targets occur at a level comparable with the spontaneous mutation rate, usually 10-5 to 10-7 per generation. Reversion (by precise excision of the IS element) is usually infrequent, with a range of rates of 10-6 to 10-10 per generation, which is 103 times less frequent than insertion.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة

الآخبار الصحية















 قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة
قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة "المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة
"المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة (نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)
(نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)