النبات

مواضيع عامة في علم النبات

الجذور - السيقان - الأوراق

النباتات الوعائية واللاوعائية

البذور (مغطاة البذور - عاريات البذور)

الطحالب

النباتات الطبية


الحيوان

مواضيع عامة في علم الحيوان

علم التشريح

التنوع الإحيائي

البايلوجيا الخلوية


الأحياء المجهرية

البكتيريا

الفطريات

الطفيليات

الفايروسات


علم الأمراض

الاورام

الامراض الوراثية

الامراض المناعية

الامراض المدارية

اضطرابات الدورة الدموية

مواضيع عامة في علم الامراض

الحشرات


التقانة الإحيائية

مواضيع عامة في التقانة الإحيائية


التقنية الحيوية المكروبية

التقنية الحيوية والميكروبات

الفعاليات الحيوية

وراثة الاحياء المجهرية

تصنيف الاحياء المجهرية

الاحياء المجهرية في الطبيعة

أيض الاجهاد

التقنية الحيوية والبيئة

التقنية الحيوية والطب

التقنية الحيوية والزراعة

التقنية الحيوية والصناعة

التقنية الحيوية والطاقة

البحار والطحالب الصغيرة

عزل البروتين

هندسة الجينات


التقنية الحياتية النانوية

مفاهيم التقنية الحيوية النانوية

التراكيب النانوية والمجاهر المستخدمة في رؤيتها

تصنيع وتخليق المواد النانوية

تطبيقات التقنية النانوية والحيوية النانوية

الرقائق والمتحسسات الحيوية

المصفوفات المجهرية وحاسوب الدنا

اللقاحات

البيئة والتلوث


علم الأجنة

اعضاء التكاثر وتشكل الاعراس

الاخصاب

التشطر

العصيبة وتشكل الجسيدات

تشكل اللواحق الجنينية

تكون المعيدة وظهور الطبقات الجنينية

مقدمة لعلم الاجنة


الأحياء الجزيئي

مواضيع عامة في الاحياء الجزيئي


علم وظائف الأعضاء


الغدد

مواضيع عامة في الغدد

الغدد الصم و هرموناتها

الجسم تحت السريري

الغدة النخامية

الغدة الكظرية

الغدة التناسلية

الغدة الدرقية والجار الدرقية

الغدة البنكرياسية

الغدة الصنوبرية

مواضيع عامة في علم وظائف الاعضاء

الخلية الحيوانية

الجهاز العصبي

أعضاء الحس

الجهاز العضلي

السوائل الجسمية

الجهاز الدوري والليمف

الجهاز التنفسي

الجهاز الهضمي

الجهاز البولي


المضادات الميكروبية

مواضيع عامة في المضادات الميكروبية

مضادات البكتيريا

مضادات الفطريات

مضادات الطفيليات

مضادات الفايروسات

علم الخلية

الوراثة

الأحياء العامة

المناعة

التحليلات المرضية

الكيمياء الحيوية

مواضيع متنوعة أخرى

الانزيمات
The ColE1 Compatibility System Is Controlled by an RNA Regulator
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المصدر:
LEWIN’S GENES XII
الجزء والصفحة:
8-4-2021
2577
The ColE1 Compatibility System Is Controlled by an RNA Regulator
KEY CONCEPTS
- Replication of ColE1 requires transcription to pass through the origin, where the transcript is cleaved by RNase H to generate a primer end.
- The regulator RNA I is a short antisense RNA that pairs with the transcript and prevents the cleavage that generates the priming end.
- The Rom protein enhances pairing between RNA I and the transcript.
The best characterized copy number and incompatibility system is that of the plasmid ColE1, a multicopy plasmid that is maintained at a steady level of about 20 copies per E. coli cell. The system for maintaining the copy number depends on the mechanism for initiating replication at the ColE1 origin, as illustrated in FIGURE 1.
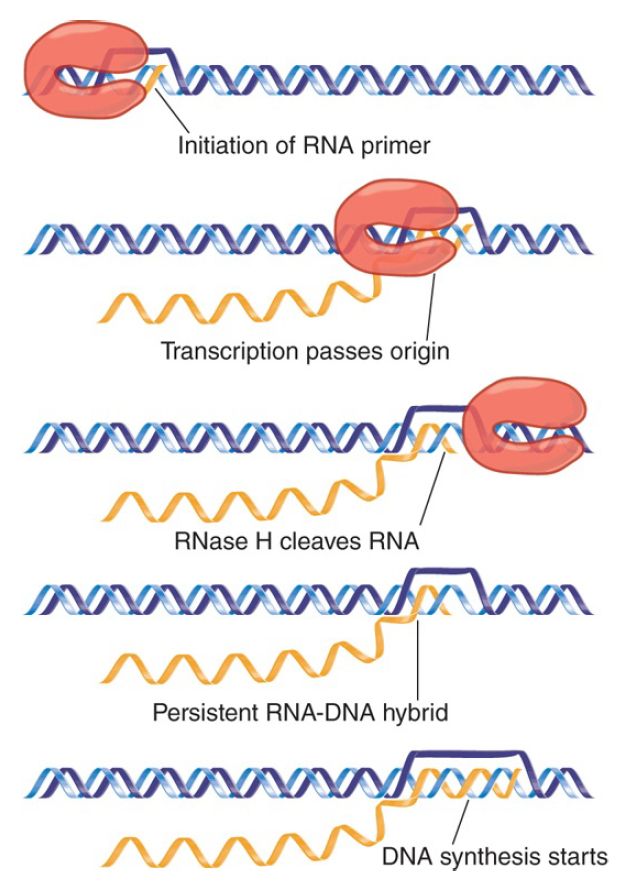
FIGURE 1. Replication of ColE1 DNA is initiated by cleaving the primer RNA to generate a 3′–OH end. The primer forms a persistent hybrid in the origin region.
Replication starts with the transcription of an RNA that initiates 555 bp upstream of the origin. Transcription continues through the origin. The enzyme RNase H (whose name reflects its specificity for a substrate of RNA hybridized with DNA) cleaves the transcript at the origin. This generates a 3′–OH end that is used as the “primer” at which DNA synthesis is initiated . The primer RNA forms a persistent hybrid with the DNA. Pairing between the RNA and DNA occurs just upstream of the origin (around position −20) and also farther upstream (around position −265).
Two regulatory systems exert their effects on the RNA primer. One involves synthesis of an RNA complementary to the primer; the other involves a protein encoded by a nearby locus.
The regulatory species RNA I is a molecule of about 108 bases and is encoded by the opposite strand from that specifying primer RNA. The relationship between the primer RNA and RNA I is illustrated in FIGURE 2. The RNA I molecule is initiated within the primer region and terminates close to the site where the primer RNA initiates. Thus, RNA I is complementary to the 5′–terminal region of the primer RNA. Base pairing between the two RNAs controls the availability of the primer RNA to initiate a cycle of replication.
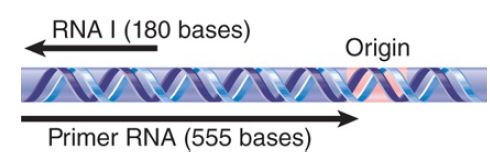
FIGURE 2. The sequence of RNA I is complementary to the 5′ region of primer RNA.
An RNA molecule such as RNA I that functions by virtue of its complementarity with another RNA encoded in the same region is called a countertranscript. This type of mechanism is another example of the use of antisense RNA .
Mutations that reduce or eliminate incompatibility between plasmids can be obtained by selecting plasmids of the same group for their ability to coexist. Incompatibility mutations in ColE1 map in the region of overlap between RNA I and primer RNA. This region is represented in two different RNAs, so either or both might be involved in the effect.
When RNA I is added to a system for replicating ColE1 DNA in vitro, it inhibits the formation of active primer RNA. The presence of RNA I, however, does not inhibit the initiation or elongation of primer RNA synthesis. This suggests that RNA I prevents RNase H from generating the 3′ end of the primer RNA. The basis for this effect lies in base pairing between RNA I and primer RNA.
Both RNA molecules have the same potential secondary structure in this region, with three duplex hairpins terminating in singlestranded loops. Mutations reducing incompatibility are located in these loops, which suggests that the initial step in base pairing between RNA I and primer RNA is contact between the unpaired loops.
How does pairing with RNA I prevent cleavage to form primer RNA? A model is illustrated in FIGURE 3. In the absence of RNA I, the primer RNA forms its own secondary structure (involving loops and stems). When RNA I is present, though, the two molecules pair and become completely double-stranded for the entire length of RNA I. The new secondary structure prevents the formation of the primer, probably by affecting the ability of the RNA to form the persistent hybrid.
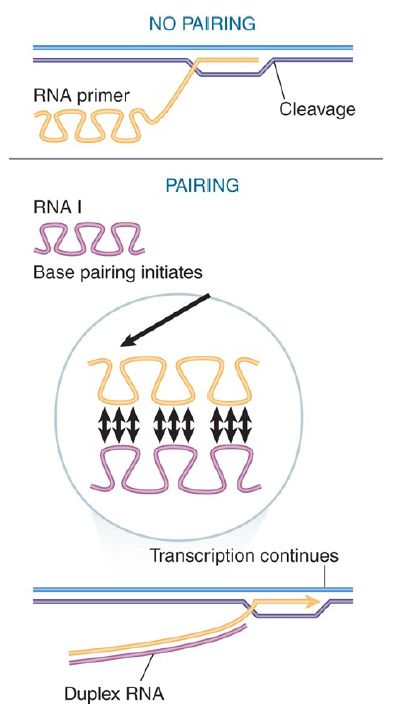
FIGURE 3. Base pairing with RNA I may change the secondary structure of the primer RNA sequence and thus prevent cleavage from generating a 3′–OH end.
The model resembles the mechanism involved in attenuation of transcription, in which the alternative pairings of an RNA sequence permit or prevent formation of the secondary structure needed for termination by RNA polymerase . The action of RNA I is exercised by its ability to affect distant regions of the primer precursor.
Formally, the model is equivalent to postulating a control circuit involving two RNA species. A large RNA primer precursor is a positive regulator and is needed to initiate replication. The small RNA I is a negative regulator that is able to inhibit the action of the positive regulator.
In its ability to act on any plasmid present in the cell, RNA I provides a repressor that prevents newly introduced DNA from functioning. This is analogous to the role of the lambda lysogenic repressor . Instead of a repressor protein that binds the new DNA, an RNA binds the newly synthesized precursor to the RNA primer.
Binding between RNA I and primer RNA can be influenced by the Rom protein, which is coded by a gene located downstream of the origin. Rom enhances binding between RNA I and primer RNA transcripts of more than 200 bases. The result is to inhibit formation of the primer.
How do mutations in the RNAs affect incompatibility? FIGURE 4 shows the situation when a cell contains two types of RNA I/primer RNA sequence. The RNA I and primer RNA made from each type of genome can interact, but RNA I from one genome does not interact with primer RNA from the other genome. This situation would arise when a mutation in the region that is common to RNA I and primer RNA occurred at a location involved in the base pairing between them. Each RNA I would continue to pair with the primer RNA encoded by the same plasmid, but might be unable to pair with the primer RNA coded by the other plasmid. This would cause the original and the mutant plasmids to behave as members of different compatibility groups.

FIGURE 4. Mutations in the region coding for RNA I and the primer precursor need not affect their ability to pair, but they may prevent pairing with the complementary RNA encoded by a different plasmid.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة

الآخبار الصحية















 قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة
قسم الشؤون الفكرية يصدر كتاباً يوثق تاريخ السدانة في العتبة العباسية المقدسة "المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة
"المهمة".. إصدار قصصي يوثّق القصص الفائزة في مسابقة فتوى الدفاع المقدسة للقصة القصيرة (نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)
(نوافذ).. إصدار أدبي يوثق القصص الفائزة في مسابقة الإمام العسكري (عليه السلام)