
Priming Is Required to Start DNA Synthesis
 المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
المؤلف:
JOCELYN E. KREBS, ELLIOTT S. GOLDSTEIN and STEPHEN T. KILPATRICK
 المصدر:
LEWIN’S GENES XII
المصدر:
LEWIN’S GENES XII
 الجزء والصفحة:
الجزء والصفحة:
 5-4-2021
5-4-2021
 3549
3549
Priming Is Required to Start DNA Synthesis
KEY CONCEPTS
- All DNA polymerases require a 3′–OH priming end to initiate DNA synthesis.
- The priming end can be provided by an RNA primer, a nick in DNA, or a priming protein.
- For DNA replication, a special RNA polymerase called a primase synthesizes an RNA chain that provides the priming end.
- E. coli has two types of priming reaction, which occur at the bacterial origin (oriC) and the Ф 174 origin.
- Priming of replication on double-stranded DNA always requires a replicase, SSB, and primase.
- DnaB is the helicase that unwinds DNA for replication in E. coli.
A common feature of all DNA polymerases is that they cannot initiate synthesis of a chain of DNA de novo, but can only elongate a chain. FIGURE 1 shows the features required for initiation. Synthesis of the new strand can start only from a preexisting 3′–OH end, and the template strand must be converted to a singlestranded condition.
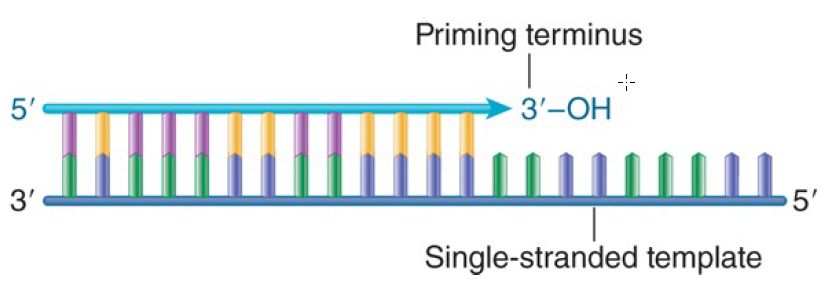
FIGURE 1. A DNA polymerase requires a 3′–OH end to initiate replication.
The 3′–OH end is called a primer. The primer can take various forms (see also FIGURE 2, which summarizes the types of priming reaction):
- A sequence of RNA is synthesized on the template, so that the free 3′–OH end of the RNA chain is extended by the DNA polymerase. This is commonly used in replication of cellular DNA and by some viruses.
- A preformed RNA (often a tRNA) pairs with the template, allowing its 3′–OH end to be used to prime DNA synthesis. This mechanism is used by retroviruses to prime reverse transcription of RNA .
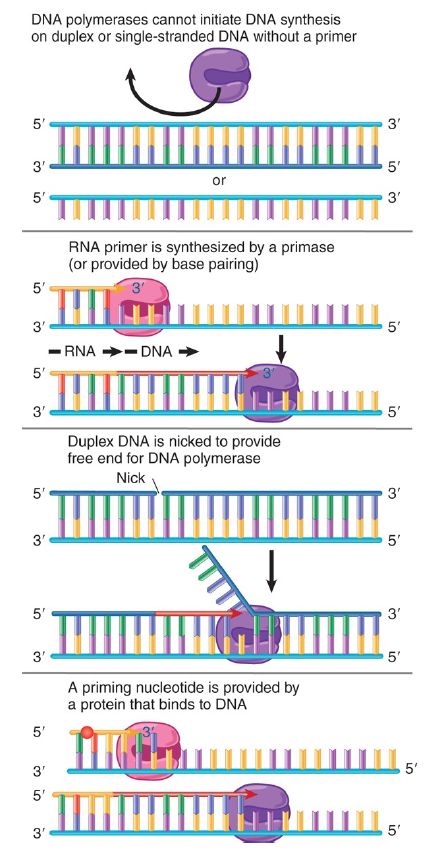
FIGURE 2 There are several methods for providing the free 3′–OH end that DNA polymerases require to initiate DNA synthesis.
- A primer terminus is generated within duplex DNA. The most common mechanism is the introduction of a nick, as used to initiate rolling circle replication. In this case, the preexisting strand is displaced by new synthesis.
- A protein primes the reaction directly by presenting a nucleotide to the DNA polymerase. This reaction is used by certain viruses . Priming activity is required to provide 3′–OH ends to start off the DNA chains on both the leading and lagging strands. The leading strand requires only one such initiation event, which occurs at the origin. There must be a series of initiation events on the lagging strand, though, because each Okazaki fragment requires its own start de novo. Each Okazaki fragment begins with a primer sequence of RNA approximately 10 bases long that provides the 3′–OH end for extension by DNA polymerase.
A primase is required to catalyze the actual priming reaction. In E. coli, this is provided by a special RNA polymerase activity, the product of the dnaG gene. The enzyme is a single polypeptide of 60 kD (much smaller than the RNA polymerase used for transcription). The primase is an RNA polymerase that is used only under specific circumstances; that is, to synthesize short stretches of RNA that are used as primers for DNA synthesis. DnaG primase associates transiently with the replication complex, and typically synthesizes a primer of approximately 10 bases. Primers begin with the sequence pppAG positioned opposite the sequence 3′–GTC-5′ in the template.
There are two types of priming reaction in E. coli:
- The oriC system, named for the bacterial origin, basically involves the association of the DnaG primase with the protein complex at the replication fork.
- The Φ X system, named originally for phage Φ X174, requires an initiation complex consisting of additional components, called the primosome. This system is used when damage causes the replication fork to collapse and it must be restarted.
At times, replicons are referred to as being of the Φ X or oriC type. The types of activities involved in the initiation reaction are summarized in FIGURE 3. Although other replicons in E. coli might have alternatives for some of these particular proteins, the same general types of activity are required in every case. A helicase is required to generate single strands, a single-strand binding protein is required to maintain the single-stranded state, and the primase synthesizes the RNA primer.
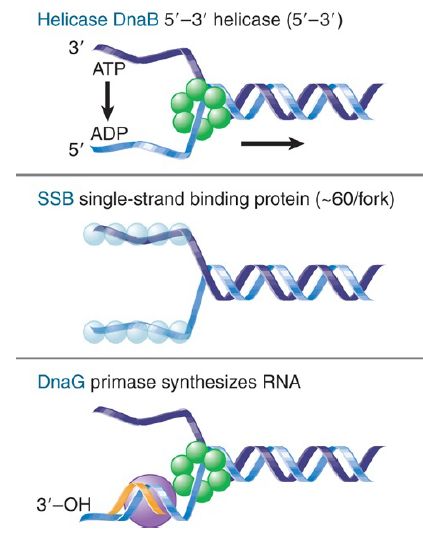
FIGURE .3 Initiation requires several enzymatic activities, including helicases, single-strand binding proteins, and synthesis of the primer.
DnaB is the central component in both Φ X and oriC replicas. It provides the 5′–3′ helicase activity that unwinds DNA. Energy for the reaction is provided by cleavage of ATP. Basically, DnaB is the active component required to advance the replication fork. In oriC replicons, DnaB is initially loaded at the origin as part of a large complex . It forms the growing point at which the DNA strands are separated as the replication fork advances. It is part of the DNA polymerase complex and interacts with the DnaG primase to initiate synthesis of each Okazaki fragment on the lagging strand.
 الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
الاكثر قراءة في مواضيع عامة في الاحياء الجزيئي
 اخر الاخبار
اخر الاخبار
اخبار العتبة العباسية المقدسة


